みなさんこんにちは!このブログでは主に
の4つのトピックについて発信しています。
今回はpythonで綺麗な散布図を描く方法の第6回です!2次元の散布図上の点の密度をカラーで表示する「密度カラー散布図」の描画方法を具体的な実装例でご紹介します。今回は特別編として、任意の軸のスケールを設定した上で、密度カラー散布図と同時にx, y各軸の1次元ヒストグラムをプロットする方法を紹介します!
この記事を読めば、軸のスケールを設定した上で1次元のヒストグラムと同時に、pythonで綺麗な2次元散布図をプロットする方法を知ることができます!ぜひ最後までご覧ください。

この記事はこんな人のお悩み解決に役立ちます!
- Pythonの作図で綺麗な散布図を作りたいけど、どうしたら良い?
- y軸を対数スケールにして散布図を作りたい
- 負の値を含むデータだけど軸を対数スケールで散布図にしたい
- Abstract | 様々なデータの散布図をヒストグラムと同時プロット
- Background | 綺麗な散布図「密度カラー散布図」とその描画方法
- Method | 軸のスケールを設定して密度カラー散布図を1次元ヒストグラムと同時に描画する実装例
- Jupyter Notebookの用意
- ライブラリのインポート
- 関数の準備
- 密度カラー散布図を実際に描画する
- Linearスケールの密度カラー散布図
- 方法1(Linearスケール: 2次元ヒストグラム)
- 方法2(Linearスケール: 密度カラー散布図(ヒストグラム・スムージングなし・線形補間))
- 方法3(Linearスケール: 密度カラー散布図(ヒストグラム・スムージングなし・Digitize))
- 方法4(Linearスケール: 2次元ヒストグラム(スムージングあり))
- 方法5(Linearスケール: 密度カラー散布図(ヒストグラム・畳み込みスムージング・線形補間))
- 方法6(Linearスケール: 密度カラー散布図(ヒストグラム・畳み込みスムージング・Digitize))
- 方法7(Linearスケール: 密度カラー散布図(ヒストグラム・KDE))
- 方法8(Linearスケール: 密度カラー散布図(KDE))
- 方法1から方法8までのプロット結果(Linearスケール)
- Logスケールの密度カラー散布図
- 方法1(Logスケール: 2次元ヒストグラム)
- 方法2(Logスケール: 密度カラー散布図(ヒストグラム・スムージングなし・線形補間))
- 方法3(Logスケール: 密度カラー散布図(ヒストグラム・スムージングなし・Digitize))
- 方法4(Logスケール: 2次元ヒストグラム(スムージングあり))
- 方法5(Logスケール: 密度カラー散布図(ヒストグラム・畳み込みスムージング・線形補間))
- 方法6(Logスケール: 密度カラー散布図(ヒストグラム・畳み込みスムージング・Digitize))
- 方法7(Logスケール: 密度カラー散布図(ヒストグラム・KDE))
- 方法8(Logスケール: 密度カラー散布図(KDE))
- 方法1から方法8までのプロット結果(Logスケール)
- Log-Modulusスケールの密度カラー散布図
- Log-Modulusスケールの定義
- プロットするデータの作成
- 方法1(Log-Modulusスケール: 2次元ヒストグラム)
- 方法2(Log-Modulusスケール: 密度カラー散布図(ヒストグラム・スムージングなし・線形補間))
- 方法3(Log-Modulusスケール: 密度カラー散布図(ヒストグラム・スムージングなし・Digitize))
- 方法4(Log-Modulusスケール: 2次元ヒストグラム(スムージングあり))
- 方法5(Log-Modulusスケール: 密度カラー散布図(ヒストグラム・畳み込みスムージング・線形補間))
- 方法6(Log-Modulusスケール: 密度カラー散布図(ヒストグラム・畳み込みスムージング・Digitize))
- 方法7(Log-Modulusスケール: 密度カラー散布図(ヒストグラム・KDE))
- 方法8(Log-Modulusスケール: 密度カラー散布図(KDE))
- 方法1から方法8までのプロット結果(Log-Modulusスケール)
- Linearスケールの密度カラー散布図
- Result | Pythonのサンプルコード
- Conclusion | まとめ
- References | 本シリーズの関連記事
Abstract | 様々なデータの散布図をヒストグラムと同時プロット
x軸やy軸のスケールを変更できるようにすることで、さまざまなデータを密度カラー散布図にプロットできます。密度カラー散布図にプロットするデータによっては、線形ではなく、対数(log)スケールなど、軸のスケールを適切なものに設定する必要があります。さらに、データ点の分布を2次元で示す密度カラー散布図を1次元のヒストグラムと同時に描画することで、データ点の分布の様子がよりわかりやすくなります。
今回の記事では、x軸やy軸のスケールを変更した上で、x軸とy軸について1次元のヒストグラムを作りながら密度カラー散布図を作成する方法を、実際のコードの書き方の例で解説します。一連の処理を関数としてまとめるので、高度な処理も簡単に実装することができます。解説するソースコードをimportしたりコピー&ペーストしてすぐに使うこともできます!ぜひ本記事を最後までお読みください(長いですが)!
Background | 綺麗な散布図「密度カラー散布図」とその描画方法
まずは密度カラー散布図についておさらいしておきましょう。この記事は密度カラー散布図シリーズの第5回です。これまでに、
- Matplotlib | Pythonで綺麗な2次元散布図を描く方法(1. 概要編)
- Matplotlib | Pythonで綺麗な2次元散布図を描く方法(2. 理論編)
- Matplotlib | Pythonで綺麗な2次元散布図の実装方法とコードの書き方(3. 基礎編)
- Matplotlib | Pythonで散布図を1次元ヒストグラムと同時にプロットする方法(Python散布図 4. 応用編)
- Matplotlib | 任意の軸スケールで密度カラー散布図をプロットする方法(Python散布図 5. 発展編)
という記事で、密度カラー散布図の有用性と基本的な考え方や実装方法ご紹介してきました。これらの内容を簡単にまとめて、密度カラー散布図の有用性と描画方法の8つの分類を復習しましょう。
散布図は密度カラー散布図としてプロットすると見やすい
散布図を作成するとき、点と点が被って様子がわからなくなることがよくあります。点の密度をカラーで示す「密度カラー散布図」を使えば、点が密集していても分布の様子を表現することができます。
通常の散布図では点の密度が高い部分が潰れる
図1は正規分布に従うデータ点と双峰分布に従うデータ点を散布図にしたものです(詳細は過去記事「Matplotlib | Pythonで綺麗な2次元散布図を描く方法1(概要編)」参照)。図1の通常の散布図では中心部が潰れています。そのため、正規分布に従うデータと双峰分布に従うデータの区別ができません。
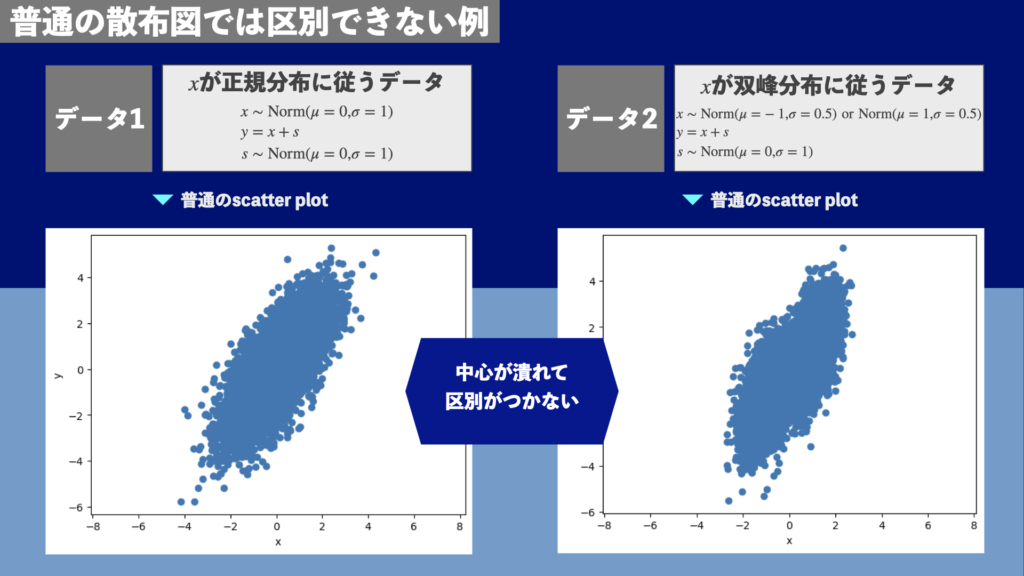
「密度カラー散布図」なら点の密度が高い部分も表現できる
一方、図2は密度カラー散布図です。点の密度をカラーで表現することで、データ点の密度が高い部分も表現できます。正規分布に従うデータ点と双峰分布に従うデータ点が明確に区別できます。データ点の密度が高い部分は情報量も多いため、このように的確に表現されるべきです。
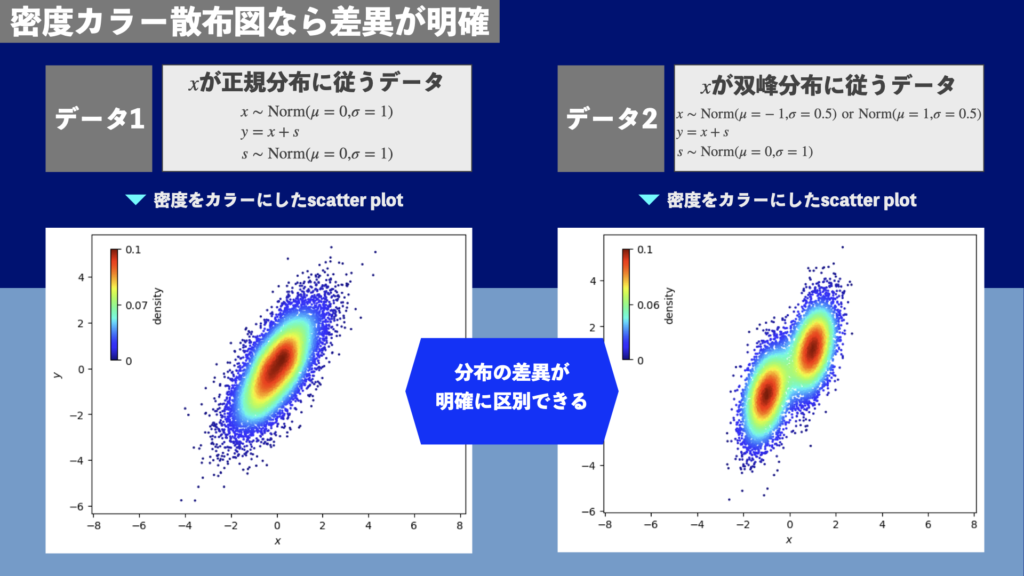
密度カラー散布図の8通りの描画方法
密度カラー散布図の描画方法は
- 密度の計算方法
- スムージングの方法
- 個々の点への密度の割当方法
によって8つに分類することができます(図3)。方法5や6が様々な場面で使いやすい描画方法です。方法1と4は散布図ではなく2次元ヒストグラムの見た目になります。方法7や8はカーネル密度推定(KDE)を使うため、データ点の数が多いと計算時間が長くなります。
今回は図3の8つの密度カラー散布図の描画方法を実装するコード例を紹介します。
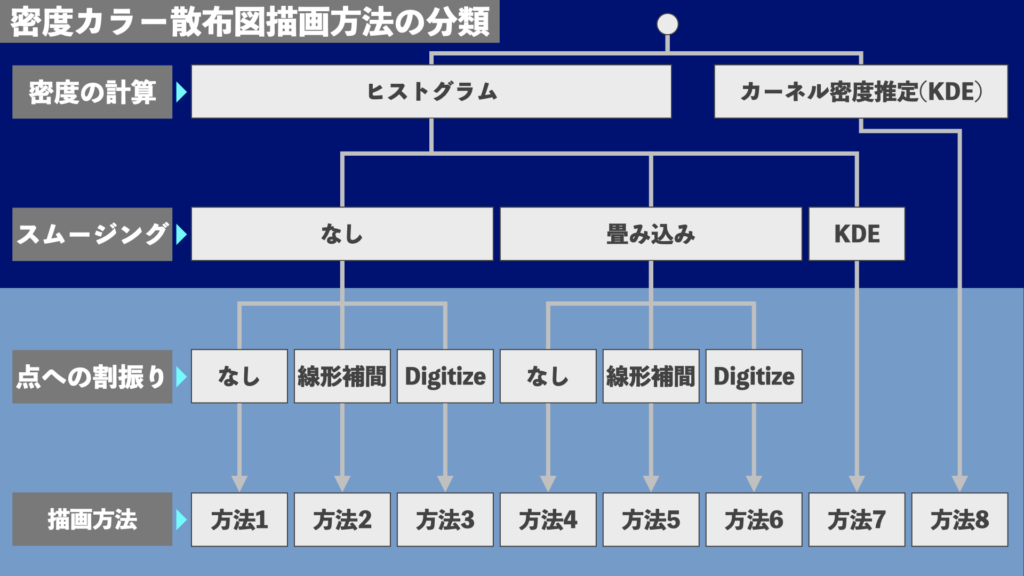
各描画方法の特徴や理論、基本的な実装方法の詳細は過去記事参照
図3の8つの描画方法の詳しい特徴や
- カーネル密度推定(KDE)
- 線形補間
- Digitize
などの用語や理論的な背景については過去記事
で解説しています。こちらもぜひご覧ください!
密度カラー散布図を単独で描画する基本的な実装方法についても過去記事
で解説していますので、こちらもご覧ください!
軸のスケール設定の必要性
密度カラー散布図にプロットするデータによっては、線形ではなく、対数(log)スケールなど、軸のスケールを適切なものに設定する必要があります。変数が正規分布に近い場合は線形スケールのヒストグラムで表現できる一方で、変数が対数正規分布に近い場合には線形スケールでは潰れて見えないためlogスケールを取る必要があります。さらに、対数スケールのデータの変化などは負の値も表現できる対数が必要になります。軸のスケール設定の必要性について、詳しくは過去記事
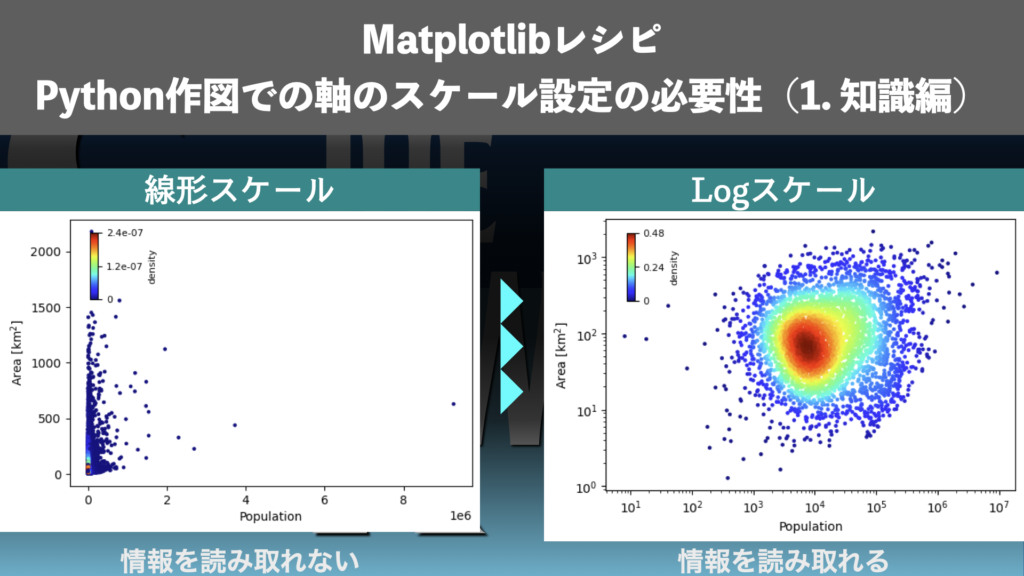
で解説していますので、そちらもご覧ください!
密度カラー散布図を1次元ヒストグラムと同時にプロット
データ点の分布を2次元で示す密度カラー散布図を1次元のヒストグラムと同時に描画することで、データ点の分布の様子がよりわかりやすくなります(図5)。今回は、x軸やy軸のスケールを任意に設定できるようにした上で、x軸とy軸に1次元ヒストグラムを追加して描画する方法を紹介します。
密度カラー散布図と1次元のヒストグラムを同時に描画する方法については過去記事
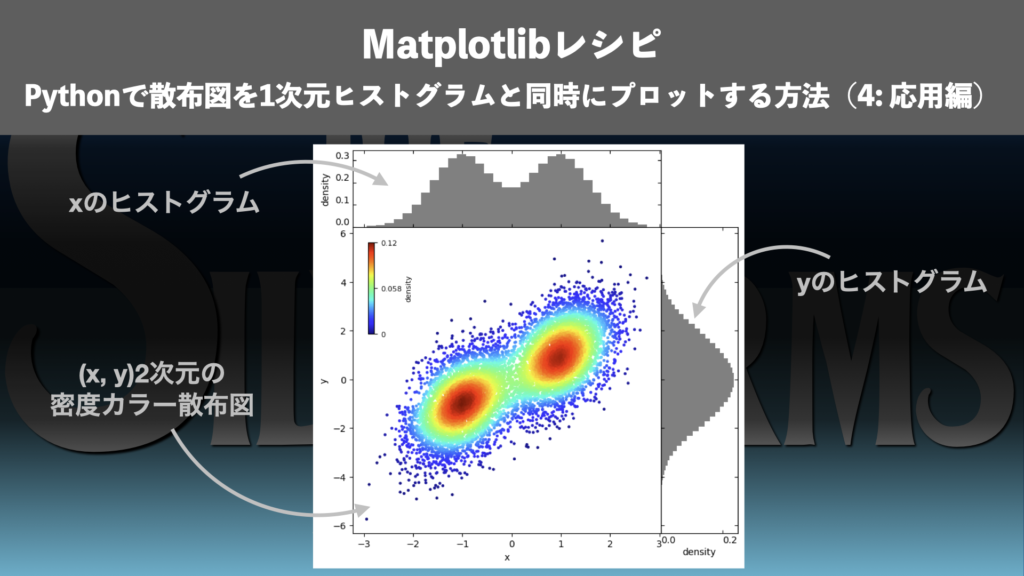
もご覧ください!
Method | 軸のスケールを設定して密度カラー散布図を1次元ヒストグラムと同時に描画する実装例
任意の軸のスケールを設定した上で、密度カラー散布図を1次元ヒストグラムと同時に描画する方法をご紹介します。具体的なコードの書き方を一つ一つ解説していきます。
ここからはJupyter Notebookで一緒に作業することを想定し、一つ一つご紹介していきます。実際にはここで用いた関数を通常のPythonソースコードにコピーして使ったり、Pythonのファイルにまとめてimportして使うのが良いでしょう。ここでは簡単のためにJupyter Notebookで紹介します。
Resultの章にJupyter Notebookでの実装例を掲載するので、そちらを参照していただいても構いません。この章では、簡単な解説とともにソースコードを一つずつ提示します。解説が不要な方はResultの章に飛んでいただき、Jupyter Notebookでの実装例を参考にする方が早いでしょう。
Jupyter Notebookの用意
まずはJupyter Notebookのファイルを開きましょう。Jupyter Notebookの使い方については過去記事「python入門講座|pythonを使ってみよう2(Jupyter Notebookを使う方法)[第5回]」もご覧ください!
ライブラリのインポート
まずはライブラリをインポートしておきます。NumPy, Scipy, Matplotlibなどを使います。以下を実行してください。
import time
import numpy as np
import scipy as sp
from scipy import stats
from scipy import interpolate
import matplotlib as mpl
import matplotlib.pyplot as plt
import matplotlib.gridspec as gridspec
import mpl_toolkits.axisartist as axisartist
import matplotlib.scale
関数の準備
密度の計算を行う関数や点への割振りを行う関数、作図する関数など、必要な関数を定義しておきます。
カラー密度散布図作成の基本的な関数
まずは以下の関数を準備しておきます。これらはカラー密度散布図作成で必ず必要となる基本関数です。
- KDEで(x, y)での密度を計算する関数:
get_density_with_KDE() - ヒストグラムで密度を計算する関数:
get_density_from_hist() - ヒストグラムを線形補間する関数:
get_interpolator_from_hist() - ヒストグラムから任意の点の密度を返す関数を作る関数:
get_func_from_histogram2d() - 一連の密度計算をまとめる関数(スケール非対応):
get_scatter_density() - カラーバーを内側に作成する関数: `draw_inset_colorbar_on_ax()`
これら6つの関数は、過去記事「Matplotlib | Pythonで綺麗な2次元散布図の実装方法とコードの書き方(3. 基礎編)」で詳しく解説しています。今回の記事ではコードを紹介するに留めて詳しい解説を省きます。詳しい解説が必要な方は上記の過去記事を御覧ください!
今回の記事では、上記6つの関数を以下の1つのコードセルで定義してしまいます。長いコードセルになってしまいますが、コピー&ペーストしやすいのでご容赦ください。以下を実行します。
# 1. get density at (x,y) using KDE with data points (x_i, y_i)
def get_density_with_KDE(
x:np.ndarray,
y:np.ndarray,
bw_method="scott",
wht_kde=None,
):
"""Get density at (x,y) using KDE with data points (x_i, y_i)
Args:
x (np.ndarray): x
y (np.ndarray): y
bw_method (str, optional): Bin width method passed to gaussian_kde. Defaults to "scott".
wht_kde (np.ndarray): weights
Returns:
v (np.ndarray): density at (x, y)
kernel (object): gaussian_kde kernel
"""
# KDE
kernel = sp.stats.gaussian_kde(
[x, y],
bw_method=bw_method,
weights=wht_kde
)
# get density
return kernel.pdf([x, y]), kernel
# 2. # get density per bin using histogram
def get_density_from_hist(
x:np.ndarray,
y:np.ndarray,
bins=["auto","auto"],
density=True
):
"""Get density per bin using histogram
Args:
x (np.ndarray): x
y (np.ndarray): y
bins (list, optional): bins passed to histogram2d. Defaults to ["auto","auto"].
density (bool, optional): If True, density is pdf (\int{\rho dx dy} = 1), otherwise number per bin
Returns:
h_vals (np.ndarray): histogram values
x/y_bins (np.ndarray): histogram bins
x/y_cens (np.ndarray): center of bins
"""
# get bins for x and y
x_bins = np.histogram_bin_edges(x, bins=bins[0])
y_bins = np.histogram_bin_edges(y, bins=bins[1])
x_cens = 0.5*(x_bins[:-1] + x_bins[1:])
y_cens = 0.5*(y_bins[:-1] + y_bins[1:])
# histogram 2d
h_tmp, *_ = np.histogram2d(x, y, bins=[x_bins, y_bins], density=density)
h_vals = h_tmp.T
# end
return h_vals, x_bins, y_bins, x_cens, y_cens
# 3. get interpolator from hist
def get_interpolator_from_hist(
x_cens:np.ndarray,
y_cens:np.ndarray,
v:np.ndarray,
fill_value=0.0,
bounds_error=False
):
"""Get interpolator
Args:
x_cens (np.ndarray): x bin center
y_cens (np.ndarray): y bin center
v (np.ndarray): histogram value
fill_value (float, optional): fill_value passed to scipy.interploate. Defaults to 0.0.
bounds_error (bool, optional): bounds_error passed to scipy.interpolate. Defaults to False.
Returns:
object: interpolation function (v = f_v([x, y]))
"""
# get interpolator
f_intrp = sp.interpolate.RegularGridInterpolator(
(x_cens, y_cens), v,
method="linear",
fill_value=fill_value,
bounds_error=bounds_error
)
# define a function
def f_dense_from_hist(xy):
"""
xy tuple of <1d array [N]>: x (xy[0]) and y (xy[1])
"""
# - get x and y - #
x, y = xy[0], xy[1]
# - get value - #
return f_intrp((x,y))
return f_dense_from_hist
# 4. get function to privide value at (x, y) from 2d histogram - #
def get_func_from_histogram2d(
x_edges:np.ndarray,
y_edges:np.ndarray,
h_vals:np.ndarray,
v_outer=[0.0, 0.0, 0.0, 0.0]
):
"""
description:
get function to provide pdf from histogram
arguments:
x_edges <1d array [n_bin_r+1]>: bin edges for r
y_edges <1d array [n_bin_rdot+1]>: bin edges for rdot
h_vals <2d array [n_bin_rdot, n_bin_r]>: histogram (pdf)
v_outer <list [4]>:
x<x_l and y<y_l: v_outer[0],
x<x_l and y>y_l: v_outer[1],
x>x_l and y<y_l: v_outer[2],
x>x_l and y>y_l: v_outer[3],
returns:
f_pdf <callable>: f_pdf(r,rdot) privides pdf
"""
# - define a function - #
def f_dense_from_hist(xy):
"""
xy tuple of <1d array [N]>: x (xy[0]) and y (xy[1])
"""
# - get x and y - #
x, y = xy[0], xy[1]
# - get index - #
i_x = np.digitize(x, x_edges)-1
i_y = np.digitize(y, y_edges)-1
idx_xy = np.where(
(0 <= i_x) & (i_x <= len(x_edges)-2) &
(0 <= i_y) & (i_y <= len(y_edges)-2)
)[0]
idx_e1 = np.where((0 <= i_x) & (i_y > len(y_edges)-2))[0]
idx_e2 = np.where((i_x > len(x_edges)-2) & (0 <= i_y))[0]
idx_e3 = np.where((i_x > len(x_edges)-2) & (i_y > len(y_edges)-2))[0]
# - get z - #
z = x * 0.0 + v_outer[0]
z[idx_xy] = h_vals[i_y[idx_xy], i_x[idx_xy]]
z[idx_e1] = v_outer[1]
z[idx_e2] = v_outer[2]
z[idx_e3] = v_outer[3]
# - return value - #
return z
# - end - #
return f_dense_from_hist
# 5. get scatter density
def get_scatter_density(
x:np.ndarray,
y:np.ndarray,
method_dense="hist",
method_smooth="conv",
method_points="interp",
bw_kde="scott",
bw_hist=["auto", "auto"],
dns_hist=True,
sig_smt=[2,2],
):
"""Get scatter density.
Args:
x (np.ndarray): x
y (np.ndarray): y
method_dense (str, optional): Method to calculate density. Defaults to "hist".
method_smooth (str, optional): Method for smoothing. Defaults to "conv".
method_points (str, optional): Method to get density at each point. Defaults to "interp".
bw_kde (str, optional): Bin width parameter for Gaussian KDE. Defaults to "scott".
bw_hist (list, optional): Bin width paramter for histogram. Defaults to ["auto", "auto"].
dns_hist (bool, optional): Density parameter for histogram. Defaults to True.
sig_smt (list, optional): Sigma [x, y] in smoothing. Defaults to [2,2].
Returns:
v (nd.array): density at (x,y)
f_v (object): function for density at arbitrary position
hist_vals (dict): "dense", "x_bins", "y_bins", "x_cens", "y_cens" from histogram
"""
# get density using histogram at first
h_vals, x_bins, y_bins, x_cens, y_cens = get_density_from_hist(
x, y, bw_hist, density=dns_hist
)
# calculate density
if method_dense == "kde":
# method 8: KDE
v, f_v = get_density_with_KDE(x, y, bw_kde) # v = f_v([x,y])
else:
# smoothing
if method_smooth == "kde":
# method7: hist2d + kde
x_ms, y_ms = np.meshgrid(x_cens, y_cens)
_, f_v = get_density_with_KDE(x_ms.ravel(), y_ms.ravel(), bw_kde, h_vals.ravel()) # v = f_v([x,y])
v = f_v([x, y])
else:
# apply smoothing
if (sig_smt[0] > 0 or sig_smt[1] > 0) and method_smooth == "conv":
h_vals = sp.ndimage.gaussian_filter(
h_vals, sig_smt, mode='nearest'
)
# map to points
if method_points == "interp":
# interpolate
f_v = get_interpolator_from_hist(x_cens, y_cens, h_vals.T)
v = f_v([x,y])
elif method_points == "digitize":
# digitize
f_v = get_func_from_histogram2d(x_bins, y_bins, h_vals)
v = f_v([x,y])
else:
# histogram
v, f_v = None, None
# histogram values into list
hist_vals = {
"dense": h_vals,
"x_bins": x_bins,
"y_bins": y_bins,
"x_cens": x_cens,
"y_cens": y_cens
}
# end
return v, f_v, hist_vals
# 6. draw inset color bar
def draw_inset_colorbar_on_ax(
ax:object,
ax_sct:object,
prm_clrbar={
'show':True,
'label':None,
'pos':[0.2,0.10,0.6,0.02],
'ori':'horizontal',
'fmt':'%.3g',
'n_ticks': 3,
'fs':10
}
):
"""Draw inset colorbar on ax
Args:
ax (object): Matplotlib axis object
ax_sct (object): Matplotlib PathCollection object returned from plt.scatter
prm_clrbar (dict, optional): Colorbar parameter. Defaults to { 'show':True, 'pos':[0.2,0.10,0.6,0.02], 'ori':'horizontal', 'fmt':'%.3g', 'n_ticks': 3, 'label':None, 'fs':10 }.
Returns:
cbar_ax (object): object returned from ax.inset_axes.
"""
# inset colorbar
if prm_clrbar['show']:
# get inset colorbar axis
cbar_ax = ax.inset_axes(prm_clrbar['pos'])
cbs = plt.colorbar(
ax_sct, cax=cbar_ax, orientation=prm_clrbar['ori'], format=prm_clrbar['fmt']
)
# ticks and ticklabels
try:
cbs.set_ticks(ax_sct.norm.inverse(np.linspace(0.0,1.0,prm_clrbar['n_ticks'])).data)
except:
print('** Warning, uninversible normalizer %s **' % (ax_sct.norm))
# label
cbs.set_label('%s' % (prm_clrbar['label']), size=prm_clrbar['fs'])
cbs.minorticks_off()
cbs.ax.tick_params(labelsize=prm_clrbar['fs'])
else:
cbar_ax = None
# end
return cbar_ax
これで基本の6つの関数が定義できました!
スケール対応が必要な関数
軸のスケール設定に対応するため, 新たに以下の関数を定義します。
- 一連の密度計算をまとめる関数(スケール対応): `get_scatter_density_scaled()`
この関数についての詳細は過去記事「Matplotlib | 任意の軸スケールで密度カラー散布図をプロットする方法(Python散布図 5. 発展編)」で解説しています。ここでは詳細な解説は行いません。
以下を実行して関数を定義します。
# get scatter density with x/y scale
def get_scatter_density_scaled(
x:np.ndarray,
y:np.ndarray,
method_dense="hist",
method_smooth="conv",
method_points="interp",
bw_kde="scott",
bw_hist=["auto", "auto"],
dns_hist=True,
sig_smt=[2,2],
x_scale="linear",
y_scale="linear"
):
"""Get scatter density.
Args:
x (np.ndarray): x
y (np.ndarray): y
method_dense (str, optional): Method to calculate density. Defaults to "hist".
method_smooth (str, optional): Method for smoothing. Defaults to "conv".
method_points (str, optional): Method to get density at each point. Defaults to "interp".
bw_kde (str, optional): Bin width parameter for Gaussian KDE. Defaults to "scott".
bw_hist (list, optional): Bin width paramter for histogram. Defaults to ["auto", "auto"].
dns_hist (bool, optional): Density parameter for histogram. Defaults to True.
sig_smt (list, optional): Sigma [x, y] in smoothing. Defaults to [2,2].
x_scale (str): scale for x. Select from mpl.scale.get_scale_names().
y_scale (str): scale for y. Select from mpl.scale.get_scale_names().
Returns:
v (nd.array): density at (x,y)
f_v (object): function for density at arbitrary position
hist_vals (dict): "dense", "x_bins", "y_bins", "x_cens", "y_cens" from histogram
v_trn (nd.array): density at transformed (x,y) for the scale
f_v_trn (object): function for density at arbitrary position at transformed (x,y) for the scale
hist_vals_trn (dict): "dense", "x_bins", "y_bins", "x_cens", "y_cens" from histogram (transformed (x,y) for the scale)
xy_trn (tuple): transformed x, y [x_trn, y_trn].
"""
# get scale & transfrm object
mpl_scl = {
"x": mpl.scale.scale_factory(x_scale, plt.subplot(111)),
"y": mpl.scale.scale_factory(y_scale, plt.subplot(111))
}
plt.close('all')
mpl_trn = {
"x": mpl_scl["x"].get_transform(),
"y": mpl_scl["y"].get_transform(),
}
mpl_inv = {
"x": mpl_trn["x"].inverted(),
"y": mpl_trn["y"].inverted()
}
# scale x, y
x_trn = mpl_trn["x"].transform(x)
y_trn = mpl_trn["y"].transform(y)
# get scatter density
v_trn, f_v_trn, hist_vals_trn = get_scatter_density(
x_trn, y_trn,
method_dense=method_dense, method_smooth=method_smooth, method_points=method_points,
bw_kde=bw_kde, bw_hist=bw_hist, dns_hist=dns_hist, sig_smt=sig_smt
)
# inverse trans form v
v = v_trn
# inverse transform f_v
def f_v(xy):
"""
xy tuple of <1d array [N]>: x (xy[0]) and y (xy[1])
"""
# transform xy
xy_trn = [
mpl_trn["x"].transform(xy[0]),
mpl_trn["y"].transform(xy[1])
]
# end
return f_v_trn(xy_trn)
# inverse transform hist_vals
hist_vals = {
"dense": hist_vals_trn["dense"],
"x_bins": mpl_inv["x"].transform(hist_vals_trn["x_bins"]),
"y_bins": mpl_inv["y"].transform(hist_vals_trn["y_bins"]),
"x_cens": mpl_inv["x"].transform(hist_vals_trn["x_cens"]),
"y_cens": mpl_inv["y"].transform(hist_vals_trn["y_cens"])
}
# transformed xy
xy_trn = [x_trn, y_trn]
# end
return v, f_v, hist_vals, v_trn, f_v_trn, hist_vals_trn, xy_trn
1次元のヒストグラムを同時に表示するための関数
1次元のヒストグラムを同時に表示するために、以下の関数を定義します。
- KDEで1次元の密度を作成する関数:
get_density1d_with_KDE() - ヒストグラムで1次元の密度を計算する関数:
get_density1d_from_hist() - 1次元の密度計算をまとめる関数:
get_density1d()
これらの関数の詳細は過去記事「Matplotlib | Pythonで散布図を1次元ヒストグラムと同時にプロットする方法(Python散布図 4. 応用編)」で解説しています。ここでは詳細な解説を省きます。
以下を実行して、上記3つの関数を定義します。
# 1. get 1d density with KDE
def get_density1d_with_KDE(
x:np.ndarray,
bw_method="scott",
wht_kde=None,
):
"""Get 1d density at x using KDE with data points x_i
Args:
x (np.ndarray): x
bw_method (str, optional): Bin width method passed to gaussian_kde. Defaults to "scott".
wht_kde (np.ndarray): weights
Returns:
v (np.ndarray): density at x
kernel (object): gaussian_kde kernel
"""
# KDE
kernel = sp.stats.gaussian_kde(
x,
bw_method=bw_method,
weights=wht_kde
)
# get density
return kernel.pdf(x), kernel
# 2. get 1d histogram with convolution
def get_density1d_from_hist(
x:np.ndarray,
bins:np.ndarray,
density=True
):
"""Get 1d histogram with convolution
Args:
x (np.ndarray): x data
bins (np.ndarray): bins for x (passed to numpy.histogram)
density (bool, optional): density passed to numpy.histogram. Defaults to True.
Returns:
np.ndarray: 1d histogram
"""
# get 1d histogram
h_vals, *_ = np.histogram(x, bins=bins, density=density)
# end
return h_vals
# 3. get 1d density
def get_density1d(
x:np.ndarray,
x_bins:np.ndarray,
x_cens:np.ndarray,
method_dense="hist",
method_smooth="conv",
bw_kde="scott",
dns_hist=True,
sig_smt=2,
):
# calculate density
if method_dense == "kde":
# method 8: KDE
_, f_v = get_density1d_with_KDE(x, bw_method=bw_kde) # v = f_v([x,y])
h_vals = f_v(x_cens)
else:
# get density from hist
h_vals = get_density1d_from_hist(
x, x_bins, density=dns_hist
)
# smoothing
if method_smooth == "kde":
# method7: hist2d + kde
_, f_v = get_density1d_with_KDE(x_cens, bw_method=bw_kde, wht_kde=h_vals) # v = f_v([x,y])
h_vals = f_v(x_cens)
else:
# apply smoothing
if sig_smt > 0 and method_smooth == "conv":
h_vals = sp.ndimage.gaussian_filter(
h_vals, sig_smt, mode='nearest'
)
# end
return h_vals
全体をplotする関数
これまでに作成した関数を取りまとめて、plotする関数を定義します。
- 全体をplotする関数: `plot_scatter_density_with_1d_scaled()`
この関数は過去記事「Matplotlib | Pythonで散布図を1次元ヒストグラムと同時にプロットする方法(Python散布図 4. 応用編)」で解説した`plot_scatter_density_with_1d`と「Matplotlib | 任意の軸スケールで密度カラー散布図をプロットする方法(Python散布図 5. 発展編)」で解説した`plot_scatter_density_scaled`を合わせたようなものになっています。ここでは詳しい説明を省略します。
以下を実行して関数を定義します。
# get scatter-density plot with 1d density
def plot_scatter_density_with_1d_scaled(
x:np.ndarray,
y:np.ndarray,
method_dense:str="hist",
method_smooth:str="conv",
method_points:str="interp",
bw_kde="scott",
bw_hist=["auto", "auto"],
dns_hist=True,
sig_smt=[2,2],
x_scale="linear",
y_scale="linear",
label_xy=["x", "y"],
cmap=plt.cm.Greens,
prm_fig={'figsize':[6*1.25,6*1.25], 'w_ratios':(4,1), 'h_ratios':(1,4)},
prm_hist1d={'color':'grey', 'fill':True, 'mrk':'+', 'ms':2},
prm_clrbar={'show':True, 'label':'density', 'pos':[0.05,0.65,0.02,0.30], 'ori':'vertical', 'fmt':'%.2g', 'n_ticks': 3, 'fs':8}
):
"""Get scatter-density plot with 1d histogram
Args:
x (np.ndarray): x
y (np.ndarray): y
method_dense (str, optional): Method to calculate density. Defaults to "hist".
method_smooth (str, optional): Method for smoothing. Defaults to "conv".
method_points (str, optional): Method to get density at each point. Defaults to "interp".
bw_kde (str, optional): Bin width parameter for Gaussian KDE. Defaults to "scott".
bw_hist (list, optional): Bin width paramter for histogram. Defaults to ["auto", "auto"].
dns_hist (bool, optional): Density parameter for histogram. Defaults to True.
sig_smt (list, optional): Sigma [x, y] in smoothing. Defaults to [2,2].
x_scale (str): scale for x. Select from mpl.scale.get_scale_names().
y_scale (str): scale for y. Select from mpl.scale.get_scale_names().
label_xy (list, optional): Label for x and y axis. Defaults to ["x", "y"].
cmap (object, optional): Colar map parameter in matplotlib. Defaults to plt.cm.Greens.
prm_fig (dict, optional): Figure parameters. Defaults to {'figsize':[6*1.25,6*1.25], 'w_ratios':(4,1), 'h_ratios':(1,4)}.
prm_hist1d (dict, optional): Parameters for 1d histogram. Defaults to {'label':'density', 'color':'grey', 'fill':True, 'mrk':'+', 'ms':2}.
prm_clrbar (dict, optional): Parameters for colar bar. Defaults to {'show':True, 'label':'density', 'pos':[0.05,0.65,0.02,0.30], 'ori':'vertical', 'fmt':'%.2g', 'n_ticks': 3, 'fs':8}.
Returns:
v (nd.array): density at (x,y)
f_v (object): function for density at arbitrary position
hist_vals (dict): "dense", "x_bins", "y_bins", "x_cens", "y_cens" from histogram
hist1d_vals (dict): "x", "y" for 1d histogram for x and y
"""
# time measurement
t_0 = time.time()
# get scatter density
(
v, f_v, hist_vals,
v_trn, f_v_trn, hist_vals_trn,
xy_trn
) = get_scatter_density_scaled(
x, y,
method_dense=method_dense,
method_smooth=method_smooth,
method_points=method_points,
bw_kde=bw_kde,
bw_hist=bw_hist,
dns_hist=dns_hist,
sig_smt=sig_smt,
x_scale=x_scale,
y_scale=y_scale
)
# get 1d histogram for transformed x/y
hist1d_vals = {}
hist1d_vals["x"] = get_density1d(
xy_trn[0], hist_vals_trn["x_bins"], hist_vals_trn["x_cens"],
method_dense=method_dense,
method_smooth=method_smooth,
bw_kde=bw_kde, #f_v.factor if method_dense == "kde" else bw_kde,
dns_hist=dns_hist,
sig_smt=sig_smt[0]
)
hist1d_vals["y"] = get_density1d(
xy_trn[1], hist_vals_trn["y_bins"], hist_vals_trn["y_cens"],
method_dense=method_dense,
method_smooth=method_smooth,
bw_kde=bw_kde, #f_v.factor if method_dense == "kde" else bw_kde,
dns_hist=dns_hist,
sig_smt=sig_smt[1]
)
# set figure
fig = plt.figure(figsize=prm_fig['figsize'])
gs = fig.add_gridspec(
2, 2,
wspace=0.0, hspace=0.0,
width_ratios=prm_fig['w_ratios'], height_ratios=prm_fig['h_ratios']
)
axs = {0:{},1:{}}
# plot 1d x hist
i1, i2 = 0, 0
axs[i1][i2] = plt.subplot(gs[i1,i2], axes_class=axisartist.Axes)
axs[i1][i2].axis['left'].major_ticklabels.set_va('bottom')
axs[i1][i2].axis['bottom'].major_ticklabels.set_visible(False)
axs[i1][i2].set_xscale(x_scale)
axs[i1][i2].set_ylabel(prm_clrbar['label'])
axs[i1][i2].stairs(
hist1d_vals["x"], edges=hist_vals["x_bins"],
color=prm_hist1d['color'], fill=prm_hist1d['fill']
)
# plot 1d y hist
i1, i2 = 1, 1
axs[i1][i2] = plt.subplot(gs[i1,i2],axes_class=axisartist.Axes)
axs[i1][i2].axis['bottom'].major_ticklabels.set_ha('left')
axs[i1][i2].axis['left'].major_ticklabels.set_visible(False)
axs[i1][i2].set_yscale(y_scale)
axs[i1][i2].set_xlabel(prm_clrbar['label'])
axs[i1][i2].stairs(
hist1d_vals["y"], edges=hist_vals["y_bins"],
color=prm_hist1d['color'], fill=prm_hist1d['fill'], orientation='horizontal'
)
# plot scatter density
i1, i2 = 1, 0
axs[i1][i2] = plt.subplot(gs[i1,i2])
axs[i1][i2].set_xlabel(label_xy[0])
axs[i1][i2].set_ylabel(label_xy[1])
axs[i1][i2].set_xscale(x_scale)
axs[i1][i2].set_yscale(y_scale)
if v is None:
ax_clr = axs[i1][i2].pcolormesh(
hist_vals["x_bins"], hist_vals["y_bins"], hist_vals["dense"],
cmap=cmap
)
else:
ax_clr = axs[i1][i2].scatter(
x, y, c=v,
cmap=cmap, s=10, lw=0.0, edgecolor='silver'
)
# inset colorbar
ax_cbar = draw_inset_colorbar_on_ax(axs[i1][i2], ax_clr, prm_clrbar=prm_clrbar)
# time measurement
t_1 = time.time()
print(' Elapsed Time: %.3g sec' % (t_1 - t_0))
# end
return v, f_v, hist_vals, hist1d_valsこれで必要な関数をすべて定義することができました!
密度カラー散布図を実際に描画する
上記で定義した一連の関数を使って、実際に密度カラー散布図を実際に描画してみましょう。
- Linear(線形)スケール
- Log(対数)スケール
- Log-Modulusスケール
の3通りについて描画していきます。
Linearスケールの密度カラー散布図
Linear(線形)スケールの密度カラー散布図を作成します。まずはデータを作成します。以下を実行してください。
# prepare data points 2
x2 = np.r_[
np.random.normal(loc=-1.0, scale=0.5, size=5000),
np.random.normal(loc=1.0, scale=0.5, size=5000)
]
y2 = x2 + np.random.normal(size=len(x2))データを作成したらプロットしていきます。方法1から方法8(図3参照)までの描画方法でプロットします。以下を順番に実行していきましょう。
方法1(Linearスケール: 2次元ヒストグラム)
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="none",
method_points="none",
cmap=plt.cm.Greens,
y_scale="linear"
)方法2(Linearスケール: 密度カラー散布図(ヒストグラム・スムージングなし・線形補間))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="none",
method_points="interp",
cmap=plt.cm.jet,
y_scale="linear"
)方法3(Linearスケール: 密度カラー散布図(ヒストグラム・スムージングなし・Digitize))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="none",
method_points="digitize",
cmap=plt.cm.jet,
y_scale="linear"
)方法4(Linearスケール: 2次元ヒストグラム(スムージングあり))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="conv",
method_points="none",
cmap=plt.cm.Greens,
y_scale="linear"
)方法5(Linearスケール: 密度カラー散布図(ヒストグラム・畳み込みスムージング・線形補間))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="conv",
method_points="interp",
cmap=plt.cm.jet,
y_scale="linear"
)方法6(Linearスケール: 密度カラー散布図(ヒストグラム・畳み込みスムージング・Digitize))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="conv",
method_points="digitize",
cmap=plt.cm.jet,
y_scale="linear"
)方法7(Linearスケール: 密度カラー散布図(ヒストグラム・KDE))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="kde",
method_points="none",
cmap=plt.cm.jet,
y_scale="linear"
)方法8(Linearスケール: 密度カラー散布図(KDE))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="kde",
method_smooth="none",
method_points="none",
cmap=plt.cm.jet,
y_scale="linear"
)方法1から方法8までのプロット結果(Linearスケール)
上記を実行すると、図6のような密度カラー散布図が表示されます。
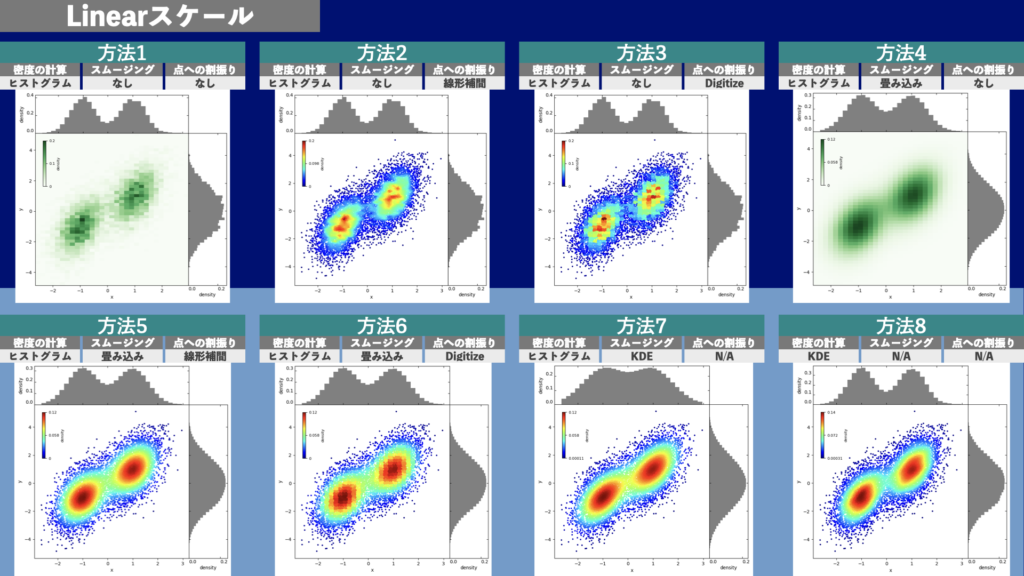
Logスケールの密度カラー散布図
次にLog(対数)スケールの密度カラー散布図を作成します。まずはデータを作成します。以下を実行してください。
# prepare data points 2
x2 = np.r_[
np.random.normal(loc=-1.0, scale=0.5, size=5000),
np.random.normal(loc=1.0, scale=0.5, size=5000)
]
y2 = 10**(x2 + np.random.normal(size=len(x2)))このデータに対して、y軸を線形のままプロットしようとするとデータ点の分布が潰れてしまい、情報を読み取ることができない図になってしまいます。上記のデータを線形でプロットした結果は過去記事「Matplotlib | 任意の軸スケールで密度カラー散布図をプロットする方法(Python散布図 5. 発展編)」で取り扱っています。過去記事もご参照ください!今回は上記のデータについて、y軸をLogスケールにしてプロットしていきます。
方法1(Logスケール: 2次元ヒストグラム)
ここからは方法1から方法8(図3参照)までの描画方法でプロットします。以下を順番に実行していきましょう。
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="none",
method_points="none",
y_scale="log",
cmap=plt.cm.Greens
)方法2(Logスケール: 密度カラー散布図(ヒストグラム・スムージングなし・線形補間))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="none",
method_points="digitize",
cmap=plt.cm.jet,
y_scale="log"
)方法3(Logスケール: 密度カラー散布図(ヒストグラム・スムージングなし・Digitize))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="none",
method_points="digitize",
cmap=plt.cm.jet,
y_scale="log"
)方法4(Logスケール: 2次元ヒストグラム(スムージングあり))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="conv",
method_points="none",
cmap=plt.cm.Greens,
y_scale="log"
)方法5(Logスケール: 密度カラー散布図(ヒストグラム・畳み込みスムージング・線形補間))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="conv",
method_points="interp",
cmap=plt.cm.jet,
y_scale="log"
)方法6(Logスケール: 密度カラー散布図(ヒストグラム・畳み込みスムージング・Digitize))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="conv",
method_points="digitize",
cmap=plt.cm.jet,
y_scale="log"
)方法7(Logスケール: 密度カラー散布図(ヒストグラム・KDE))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="kde",
method_points="none",
cmap=plt.cm.jet,
y_scale="linear"
)方法8(Logスケール: 密度カラー散布図(KDE))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="kde",
method_smooth="none",
method_points="none",
cmap=plt.cm.jet,
y_scale="linear"
)方法1から方法8までのプロット結果(Logスケール)
上記を実行すると、図7のような密度カラー散布図が表示されます。

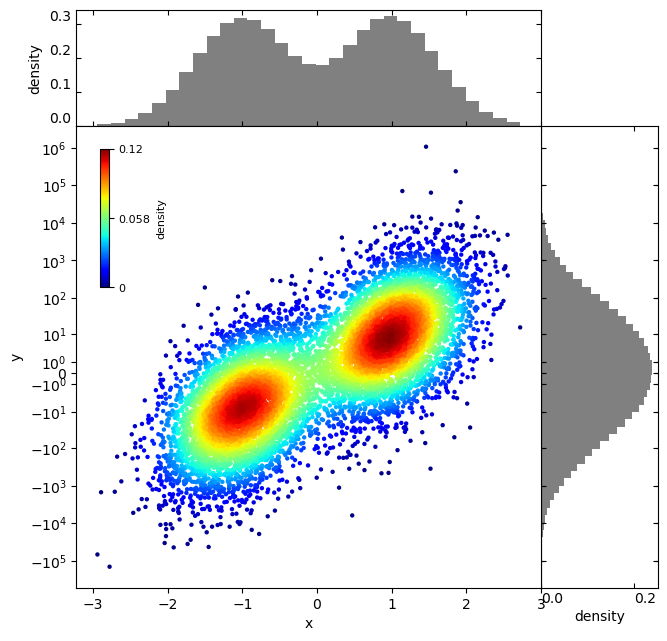
図7ではy軸のスケールが見づらいので、方法5だけを図8に表示します。
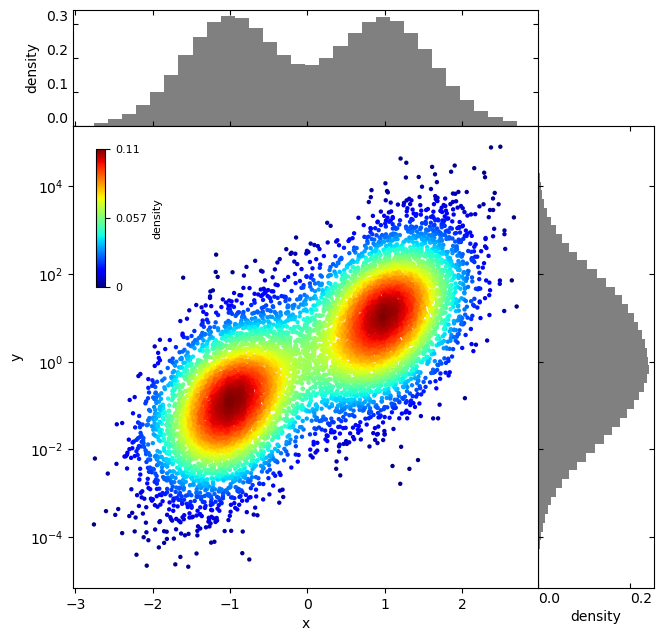
Log-Modulusスケールの密度カラー散布図
最後にLog-Modulus(線形)スケールの密度カラー散布図を作成します。
Log-Modulusスケールの定義
まずはLog-Modulusスケールをmatplotlibのscaleオブジェクトとして定義します。以下のコードセルを実行してください。なお、Matplotlibに任意のスケールを定義する方法については、過去記事「Matplotlib | 自作のスケールを定義する方法: Custom scale(Python軸スケール5)」で解説しています。詳細を知りたい方は過去記事をご覧ください。ここではコードを実行するだけに留めます。
# transformer class
class LogModulusTransform(mpl.scale.Transform):
input_dims = output_dims = 1
def __init__(self):
super().__init__()
# define parameters to set locators and formatters
self.base = 10.
self.linthresh = 1.
self.linscale = 1
self._linscale_adj = (self.linscale / (1.0 - self.base ** -1))
self._log_base = np.log(self.base)
def transform_non_affine(self, values):
return np.sign(values) * np.log10(np.fabs(values) + 1)
def inverted(self):
return InvertedLogModulusTransform()
# inverted transform class
class InvertedLogModulusTransform(mpl.scale.Transform):
input_dims = output_dims = 1
def __init__(self):
super().__init__()
# define parameters to set locators and formatters
self.base = 10
self.linthresh = 1
self.invlinthresh = 1
self.linscale = 1
self._linscale_adj = (self.linscale / (1.0 - self.base ** -1))
def transform_non_affine(self, values):
return np.sign(values) * (10**(np.fabs(values)) - 1)
def inverted(self):
return LogModulusTransform()
# scale class
class LogModulusScale(mpl.scale.ScaleBase):
"""
Log-Modulus scale converts positive and negative values
into quasi-log scale.
The scale function:
sign(x) * log(|x| + 1)
The inverse function:
sign(x) * (10**|x| - 1)
"""
# scale name
name = 'logmodulus'
def __init__(self, axis):
super().__init__(axis)
self._transform = LogModulusTransform()
def get_transform(self):
return self._transform
def set_default_locators_and_formatters(self, axis):
axis.set_major_locator(mpl.ticker.SymmetricalLogLocator(self.get_transform()))
axis.set_major_formatter(mpl.ticker.LogFormatterSciNotation(10))
axis.set_minor_locator(mpl.ticker.SymmetricalLogLocator(self.get_transform(), None))
axis.set_minor_formatter(mpl.ticker.NullFormatter())
# register scale
mpl.scale.register_scale(LogModulusScale)プロットするデータの作成
次に、データを作成します。以下を実行してください。
# prepare data points 2
x2 = np.r_[
np.random.normal(loc=-1.0, scale=0.5, size=5000),
np.random.normal(loc=1.0, scale=0.5, size=5000)
]
y_tmp = (x2 + np.random.normal(size=len(x2)))
y2 = np.sign(y_tmp) * (10**(np.fabs(y_tmp))-1)方法1(Log-Modulusスケール: 2次元ヒストグラム)
ここからは方法1から方法8(図3参照)までの描画方法でプロットします。以下を順番に実行していきましょう。
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="none",
method_points="none",
y_scale="logmodulus",
cmap=plt.cm.Greens
)方法2(Log-Modulusスケール: 密度カラー散布図(ヒストグラム・スムージングなし・線形補間))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="none",
method_points="interp",
cmap=plt.cm.jet,
y_scale="logmodulus"
)方法3(Log-Modulusスケール: 密度カラー散布図(ヒストグラム・スムージングなし・Digitize))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="none",
method_points="digitize",
cmap=plt.cm.jet,
y_scale="logmodulus"
)方法4(Log-Modulusスケール: 2次元ヒストグラム(スムージングあり))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="conv",
method_points="none",
cmap=plt.cm.Greens,
y_scale="logmodulus"
)方法5(Log-Modulusスケール: 密度カラー散布図(ヒストグラム・畳み込みスムージング・線形補間))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="conv",
method_points="interp",
cmap=plt.cm.jet,
y_scale="logmodulus"
)方法6(Log-Modulusスケール: 密度カラー散布図(ヒストグラム・畳み込みスムージング・Digitize))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="conv",
method_points="digitize",
cmap=plt.cm.jet,
y_scale="logmodulus"
)方法7(Log-Modulusスケール: 密度カラー散布図(ヒストグラム・KDE))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="hist",
method_smooth="kde",
method_points="none",
cmap=plt.cm.jet,
y_scale="logmodulus"
)方法8(Log-Modulusスケール: 密度カラー散布図(KDE))
_ = plot_scatter_density_with_1d_scaled(
x2,
y2,
method_dense="kde",
method_smooth="none",
method_points="none",
cmap=plt.cm.jet,
y_scale="logmodulus"
)方法1から方法8までのプロット結果(Log-Modulusスケール)
上記を実行すると、図9のような密度カラー散布図が表示されます。図5や図7と似たような図に見えますが、y軸をよく見るとLog-Modulusスケールになっています。
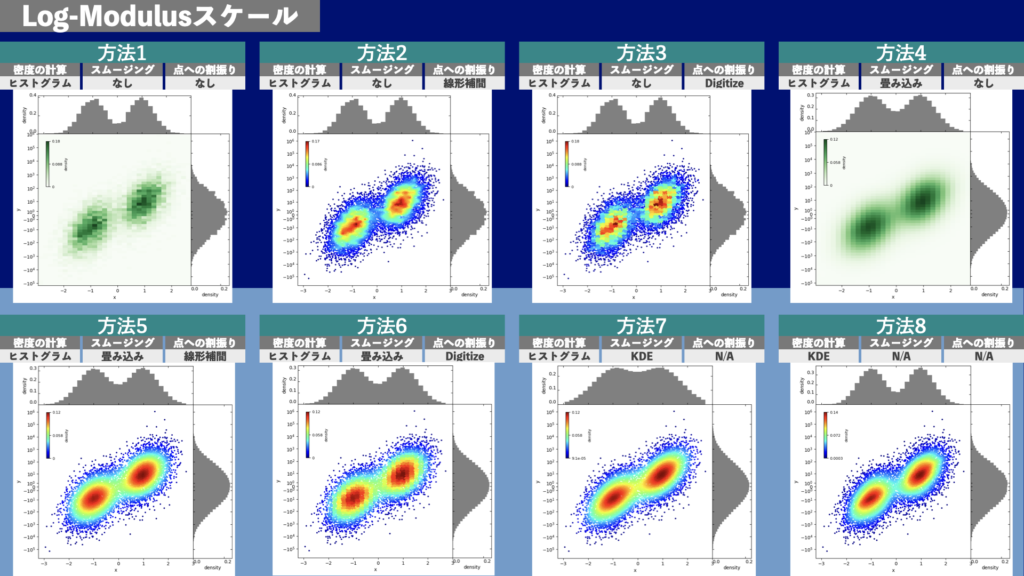
図9ではy軸のスケールが見づらいので、方法5だけを図10に表示します。
Result | Pythonのサンプルコード
今回のPythonコードの実装例を掲載します(Jupyter Notebook)。参考にどうぞ!
Conclusion | まとめ
最後までご覧頂きありがとうございます!
Pythonを使って、任意のx軸y軸スケールで1次元のヒストグラムと同時に密度カラー散布図を作成する方法をコードの実装例付きでご紹介しました!
データ点の分布を2次元で示す密度カラー散布図を1次元のヒストグラムと同時に描画することで、データ点の分布の様子がよりわかりやすくなります。さらに、x軸やy軸のスケールを変更できるようにすることで、さまざまなデータを密度カラー散布図にプロットできます。
今回のように必要な処理を関数としてまとめておくことで、このような高度な処理も簡単に実装することができます。一度関数として定義したソースコードを持っておけば、importしたりコピー&ペーストしてすぐに使うことができます!ぜひ一度、やってみてください!
以上「Matplotlib | 密度カラー散布図を任意の軸スケールで1次元ヒストグラムと同時にプロットする方法(Python散布図 6. 特別編)」でした!
またお会いしましょう!Ciao!
References | 本シリーズの関連記事
本シリーズ「Pythonで綺麗な2次元散布図を描く方法」の関連記事は以下です。 第1回の概要編に加え、密度カラー散布図の描き方の分類(2. 理論編)、基本的な実装方法(3. 基礎編)、1次元のヒストグラムと同時に描画する方法(4. 応用編)、軸のスケール設定を組み合わせる実装方法(5. 発展編)、任意の軸スケールで1次元ヒストグラムと同時に描画する方法(6.特別編)など詳しく扱っておりますので参考にどうぞ!
- Matplotlib | Pythonで綺麗な2次元散布図を描く方法(1. 概要編)
- Matplotlib | Pythonで綺麗な2次元散布図を描く方法(2. 理論編)
- Matplotlib | Pythonで綺麗な2次元散布図の実装方法(3. 基礎編)
- Matplotlib | Pythonで散布図を1次元ヒストグラムと同時にプロットする方法(4. 応用編)
- Matplotlib | 任意の軸スケールで密度カラー散布図をプロットする方法(Python散布図 5. 発展編)
- Matplotlib | 密度カラー散布図を任意の軸スケールで1次元ヒストグラムと同時にプロットする方法(Python散布図 6. 特別編)

コメント